Mehr zu Biophysik

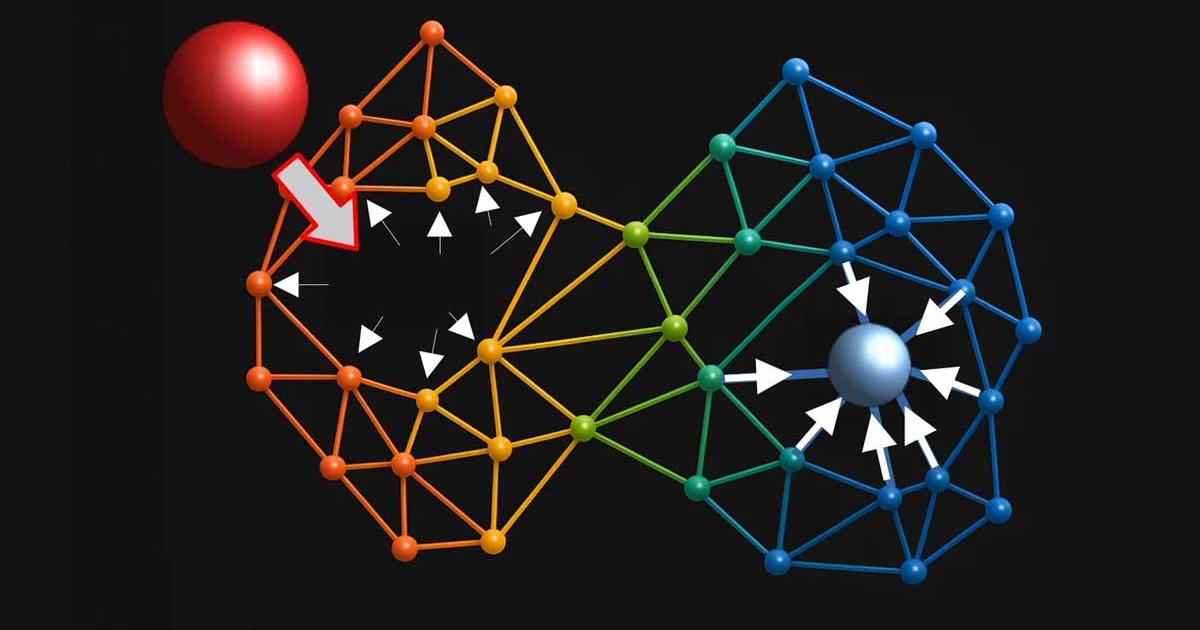
Die Forschenden untersuchten diesen Mechanismus anhand elastischer Netzwerkmodelle von Proteinen. Dabei handelt es sich um vereinfachte Darstellungen, in denen Proteine als Netzwerke aus Federn modelliert werden, die ihre Aminosäuren verbinden. Mithilfe dieser Modelle entwickelten das Team eine Methode, um zu analysieren, wie Proteinstrukturen nichtlinear auf generische mechanische Störungen reagieren, wie beispielsweise die Bindung eines Ligandenmoleküls. Sie fanden heraus, dass Störungen an Schlüsselstellen in Proteinen mit allosterischer Aktivität eine spezifische Kopplung veranlassen: Steife, lokalisierte Bewegungen sind mechanisch mit breiteren, weicheren Verformungen an anderen Stellen der Struktur verbunden. Störungen an nicht-allosterischen Stellen führen dagegen zu gleichmäßigeren und weniger gerichteten mechanischen Reaktionen. Dieser Kopplungsmechanismus erklärt, wie minimale Veränderungen an einer Stelle eines Proteins spezifische funktionelle Effekte an entfernten Stellen auslösen können. Interessanterweise sind die Aminosäuresequenzen, die für die Übertragung dieser Veränderungen verantwortlich sind, evolutionär konserviert – sie kommen also in vielen Organismen in nahezu unveränderter Form vor.
Die Ergebnisse bestätigen die Hypothese, dass Allosterie eine grundlegende Eigenschaft von Proteinstrukturen ist, die in bestimmten Fällen selektiv verstärkt wird. Diese Reaktion ist nichtlinear, gerichtet und tritt nur auf, wenn das Molekül aktiv gestört wird, beispielsweise durch die Bindung eines anderen Moleküls. Damit ist Allosterie ein grundlegend nicht im Gleichgewicht befindliches Phänomen – also ein dynamischer Prozess, der sich nicht allein durch Gleichgewicht erklärt lässt. Die neuen Erkenntnisse könnten für die Entwicklung neuer Biomoleküle mit modulierten allosterischen Eigenschaften sowie für die Wirkstoffforschung und die synthetische Biologie von Nutzen sein. [MPINAT / dre]