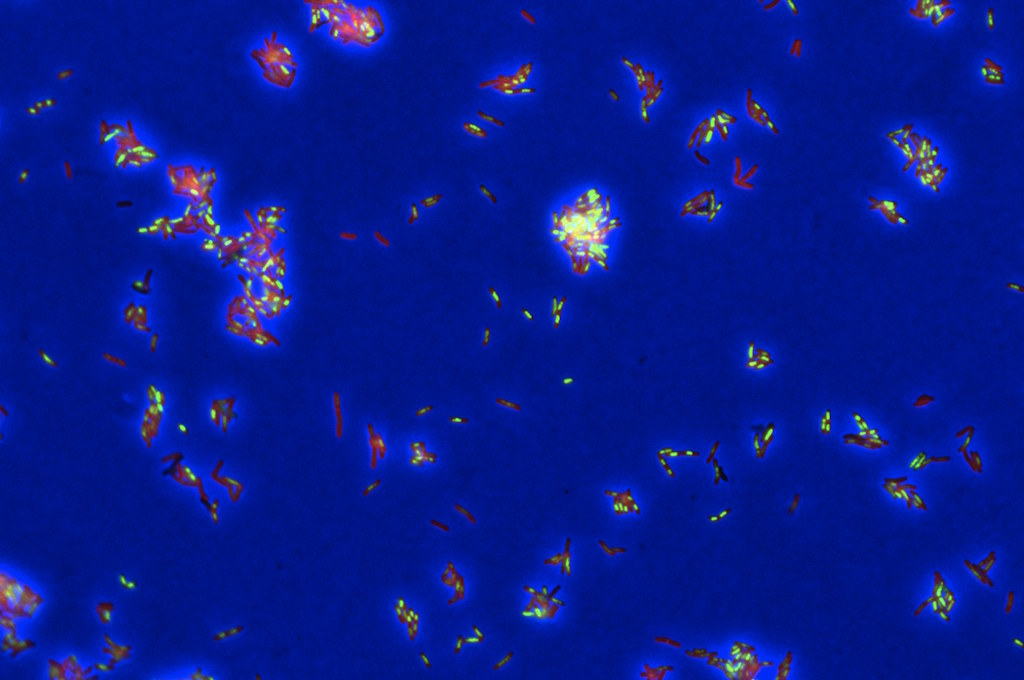
Legenda da imagem: Um “retrato da morte” de bactérias da tuberculose tratadas com um antibiótico e coradas para revelar características físicas de células individuais. Imagem: Cortesia do Laboratório Aldridge
A Tufts University apresentou uma abordagem de inteligência artificial que permite decifrar, com granularidade molecular, como os fármacos matam a bactéria da tuberculose (TB), facilitando o desenho de combinações terapêuticas mais rápidas e eficazes. O trabalho, publicado na revista Cell Systems (Johnson, W. C. et al., 2025, Integration of multi-modal measurements identifies critical mechanisms of tuberculosis drug action, Cell Systems, 16(8), 101348. DOI: https://doi.org/10.1016/j.cels.2025.101348), introduz a ferramenta DECIPHAER (decoding cross-modal information of pharmacologies via autoencoders), que integra perfis morfológicos de células bacterianas com perfis transcricionais para inferir mecanismos de morte celular.
A tuberculose continua a ser a doença infecciosa mais mortal a nível global e uma das mais difíceis de curar. O tratamento padrão exige um esquema de múltiplos fármacos durante, pelo menos, seis meses, e cerca de um em cada cinco doentes apresenta formas resistentes aos medicamentos de primeira linha. Perante este cenário, a equipa liderada por Bree Aldridge, professora de biologia molecular e microbiologia na Tufts University School of Medicine e de engenharia biomédica na Tufts University School of Engineering, desenvolveu um método que supera a limitação clássica de “ver o efeito, mas não o encadeamento causal” da ação farmacológica.
O ponto de partida foi a criação de perfis morfológicos de Mycobacterium tuberculosis: imagens de alta resolução captadas no momento da morte celular após exposição a diferentes compostos, que exibem assinaturas de dano — alterações de forma, de estrutura interna ou de integridade da parede celular — comparáveis às geradas por antibióticos com modos de ação já conhecidos. A inovação surge quando esses sinais visuais são ligados, através de modelos de IA, a leituras detalhadas da atividade génica (perfis transcricionais), permitindo associar padrões morfológicos a alterações específicas na expressão de genes bacterianos.
Segundo Bree Aldridge, esta integração acrescenta precisão à interpretação dos efeitos dos fármacos na célula e ao motivo pelo qual a bactéria morre. No processo de validação do DECIPHAER, a equipa detetou que um candidato a fármaco em desenvolvimento clínico, inicialmente presumido como inibidor da síntese da parede celular por analogia com compostos semelhantes, atua, afinal, perturbando a cadeia respiratória e a produção de energia. Esta reclassificação mecanística tem implicações diretas na seleção de combinações de fármacos, na previsão de sinergias e na mitigação de antagonismos.
Uma vantagem operacional do DECIPHAER é a capacidade de inferir o impacto molecular a partir de imagens, reduzindo a necessidade de sequenciação de RNA — um procedimento mais caro e demorado — e permitindo testar, com maior rapidez, condições de crescimento, variantes genéticas e esquemas combinatórios. A equipa planeia aplicar a ferramenta em estudos de combinações no próprio laboratório e abrir colaborações para acelerar o desenvolvimento de novos tratamentos contra a TB. A abordagem é, em princípio, extensível a outras doenças infeciosas e ao cancro.
O primeiro autor do estudo é William C. Johnson, estudante de doutoramento em microbiologia molecular na Tufts Graduate School of Biomedical Sciences. O trabalho teve apoio parcial da Fundação Gates e dos National Institutes of Health (bolsa T32AI007422). A lista completa de autores, financiadores, metodologia, limitações e potenciais conflitos de interesse encontra‑se no artigo publicado (https://doi.org/10.1016/j.cels.2025.101348).
Referência: Johnson, W. C. et al. (2025). Integration of multi-modal measurements identifies critical mechanisms of tuberculosis drug action. Cell Systems, 16(8), 101348. https://doi.org/10.1016/j.cels.2025.101348
NR/HN/AlphaGalileo